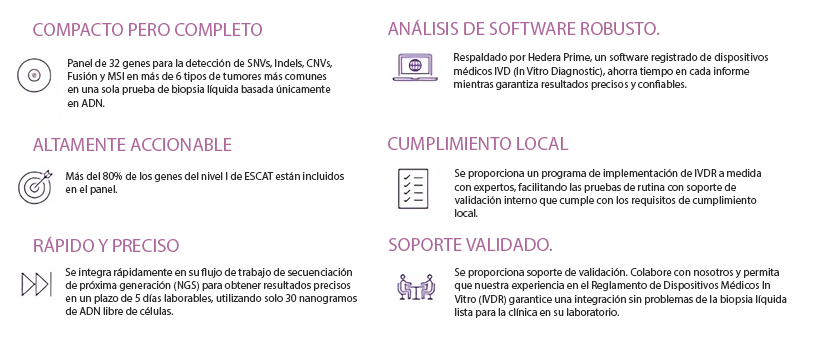
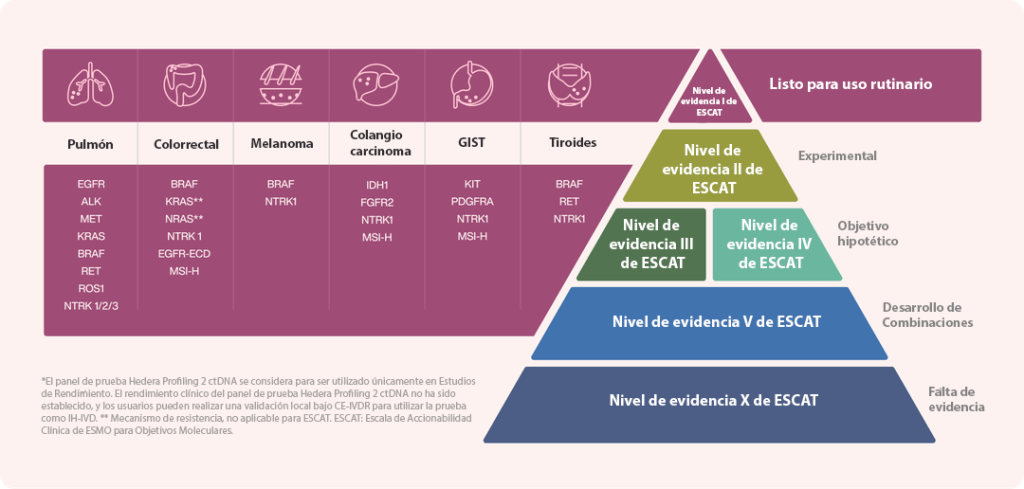
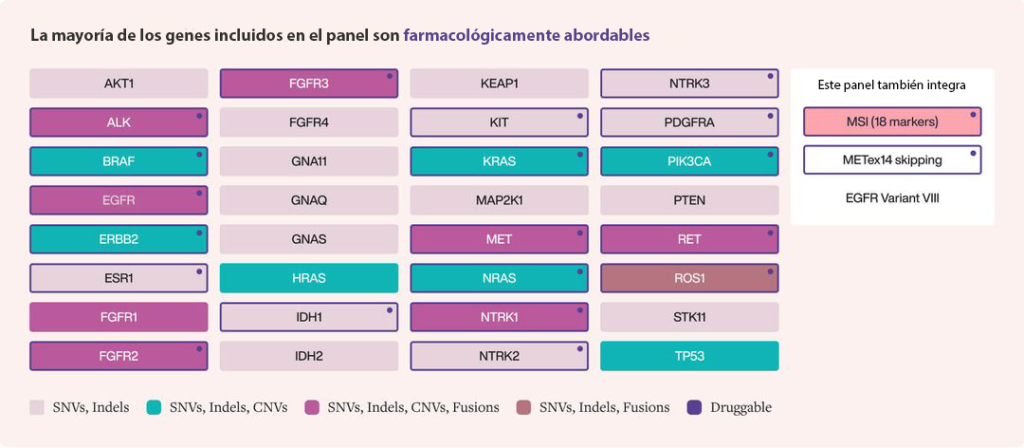
El diseño único de la prueba permite evaluar una amplia gama de biomarcadores en más de 6 tipos comunes de tumores sólidos en muestras de sangre, incluyendo variantes de un solo nucleótido (SNVs), inserciones y deleciones (Indels), ganancias y pérdidas en el número de copias (CNVs), fusiones y microsatélites inestables (MSI), todo en un flujo de trabajo de secuenciación NGS simplificado y robusto, basado únicamente en ADN. El panel consta de 32 genes, con más del 80% de todos los genes del nivel I de ESCAT incluidos hasta la fecha en las directrices de la ESMO.
- Tamaño del panel: 90 kb
- Instrumentos compatibles: Serie Illumina NextSeq y NovaSeq
- Multiplexado: hasta 6 muestras en un instrumento NextSeq 500/550 (flujo de salida Mid)
- Tamaño de la muestra: se requieren al menos 5 mL de plasma (se recomiendan tubos de colección de sangre cfDNA BCT de Streck)
- Entrada de cfDNA: mínimo de 10 ng de cfDNA, se recomiendan 30 ng de cfDNA.
- Método de preparación de la biblioteca: química de captura híbrida
- Tecnologías UMI/UDI: permiten la corrección de errores y evitan la interferencia de índices para una mayor sensibilidad y especificidad.